
이번 포스팅에서는 AI 모델을 구현할 때, 데이터 전처리 단계에서 스케일 조정을 해야하는 이유와 방법에 대해서 다룬다.
데이터 전처리가 필요한 이유?
사이킷런과 같은 머신러닝 패키지에 준비되어있는 데이터는 대부분 실습을 위한 것이므로 잘 가공되어 있다. 하지만 실전에서 수집된 데이터는 그렇지 않다. 누락된 값이 있을 수도 있고 데이터의 형태가 균일하지 않을 수도 있다. 이런 데이터들을 그대로 사용하면 제대로 결과를 얻을 수 없다. 이런 경우 데이터를 적절히 가공하는 데이터 전처리(Data preprocessing) 과정이 필요하다.
그런데, 잘 정리된 데이터에 대해서도 전처리를 해야하는 경우가 생기는데, 바로 특성의 스케일이 다른 경우이다. 스케일이란 어떤 특성이 가지고 있는 값의 범위를 말한다. 예를 들면 다음과 같은 데이터는 형태도 균일하고 누락된 값도 없지만 스케일이 다른 경우이다.
| 당도 | 무게 | |
| 사과1 | 4 | 540 |
| 사과2 | 8 | 700 |
| 사과3 | 2 | 480 |
사과의 당도 범위는 1~10이고, 무게의 범위는 500~1000이다. 바로 이런 경우 두 특성의 스케일 차이가 크다고 말한다. 어떤 알고리즘들은 스케일에 민감하여 모델의 성능에 영향을 주는데, 주로 경사하강법을 사용하는 알고리즘들이 스케일에 민감하다. 실제로 스케일을 조정한 경우와 하지 않은 경우 얼마나 차이가 나는지 확인해보자.
스케일 조정이 필요한 이유!
1. 훈련데이터 준비하고 스케일 비교하기
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
cancer = load_breast_cancer()
x = cancer.data
y = cancer.target
x_train, x_test, y_train, y_test = train_test_split(x, y, stratify=y, test_size=0.2, random_state=42)
import matplotlib.pyplot as plt
print(cancer.feature_names[[2,3]])
plt.boxplot(x_train[:,2:4])
plt.xlabel('feature')
plt.ylabel('value')
plt.show()
사이킷런에서 제공하는 유방암 데이터를 로드한 후, mean perimeter와 mean area 특성을 가져와 박스플롯을 그려 스케일을 비교해보았다.

전자는 100~200 사이에 값들이 위치한 반면에 후자는 200~2000 사이에 값들이 집중되어 있다. 이렇게 특성에 스케일이 다른 경우 경사하강법을 적용하면 가중치가 어떻게 변하는지 가중치의 변화 추이를 확인해보자.
2. 가중치 기록하고 업데이트 양 조절하기
def __init__(self) :
self.w = None
self.b = None
self.losses = []
self.w_history = []
...
# (4) 훈련메소드
def fit(self, x, y, epochs=100) :
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy()) # 초기 가중치 기록
np.random.seed(42)
for i in range(epochs) :
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes :
z = self.linear(x[i])
a = self.activation(z)
err = -(y[i] - a)
w_grad, b_grad = self.backprop(x[i], err)
self.w -= w_grad
self.b -= b_grad
self.w_history.append(self.w.copy()) # 학습 가중치 기록
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1-y[i])*np.log(1-a))
self.losses.append(loss/len(y))
가중치를 저장할 배열 w_history를 정의하고, 가중치가 학습될 때마다 copy()를 통해 가중치를 기록했다.
# 모델 학습
layer = SingleLayer()
layer.fit(x_train, y_train)
layer.score(x_test, y_test)
# 정확도 결과 : 0.9298245614035088
# 가중치 그래프 그리기
w2 = []
w3 = []
for w in layer.w_history :
w2.append(w[2])
w3.append(w[3])
plt.plot(w2,w3)
plt.plot(w2[-1], w3[-1], 'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
다음으로 새로운 모델로 학습을 진행, 정확도를 계산하고 가중치 변화 그래프를 그렸다.

그래프를 그려보니 전자에 비해 후자의 스케일이 크니 w3의 값이 학습 과정에서 큰 폭으로 흔들리며 변화하고 있다. 반면에 w2는 0부터 시작하여 조금씩 최적값에 가까워진다. 이러한 현상을 'w3에 대한 그레이디언트가 크기 때문에 w3축을 따라 가중치가 크게 요동치고 있다' 고 말한다. 즉 가중치의 최적값에 도달하는 동안 w3값이 크게 요동치므로 모델이 불안정하게 수렴한다는 것을 알 수 있다. 이런 현상을 줄일 수 있는 방법이 스케일을 조정하는 방법이다.
스케일을 조정하는 방법!
스케일을 조정하는 방법은 많지만 신경망에서 자주 사용하는 방법은 표준화(Standardization)이다. 표준화는 특성값에서 평균을 빼고 표준편차로 나누면 된다. 표준화를 하면 평균이 0이고 분산이 1인 특성이 만들어진다. 공식은 아래와 같다.

사이킷런에서 StandardScaler 클래스가 준비되어 있지만 여기서는 직접 구현해본다.
# 스케일 조정
train_mean = np.mean(x_train, axis=0)
train_std = np.std(x_train, axis=0)
x_train_scaled = (x_train - train_mean) / train_std
# 스케일 조정한 데이터 셋으로 학습 진행 및 가중치 그래프 그리기
layer2 = SingleLayer()
layer2.fit(x_train_scaled, y_train)
w2 = []
w3 = []
for w in layer2.w_history :
w2.append(w[2])
w3.append(w[3])
plt.plot(w2,w3)
plt.plot(w2[-1], w3[-1],'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
공식에 따라 스케일을 조정하고, 새로운 layer를 만들어 학습을 진행했다. 동일한 방법으로 가중치 그래프를 그렸다.

이전 그래프와는 확실히 다르다. w2와 w3의 변화 비율이 비슷하기 때문에 대각선 방향으로 가중치가 이동했다. 또한 두 특성의 스케일을 비슷하게 맞췄으므로 최적값에 빠르게 근접하고 있다.
그럼 이제 새로운 모델로 성능을 평가해보자.
layer2.score(x_test, y_test)
# 0.3726567123542321
그런데 성능이 매우 안 좋다. 이는 테스트 셋의 스케일을 바꾸지 않았기 때문이다. 모델 학습 단계에서 훈련 데이터의 스케일을 조정한 후에 가중치를 학습했기 때문에 테스트 데이터 역시 스케일을 조정해주어야한다. 그런데 이때 주의해야할 점이 하나 있다. 그것은 바로 훈련세트와 테스트세트를 동일한 비율로 스케일 조정을 해야한다는 점이다. 우선 훈련세트와 테스트세트를 다른 비율로 스케일 조정 했을 때 어떻게 되는지 확인해보자.
원본 vs 스케일 조정 후 산점도 (다른 비율로 스케일 조정)
# 원본 데이터의 산점도
plt.plot(x_train[:50,0], x_train[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()
# 테스트 데이터의 스케일 조정
test_mean = np.mean(x_test, axis=0)
test_std = np.std(x_test, axis=0)
x_test_scaled = (x_test-test_mean)/test_std
# 스케일 조정 후의 산점도
plt.plot(x_train[:50,0], x_train_scaled[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test_scaled[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()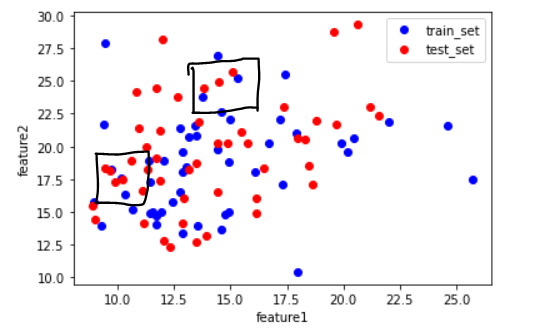
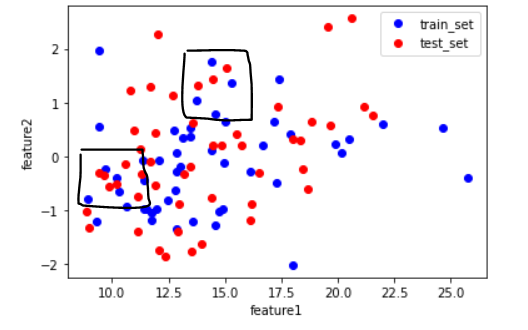
테스트 데이터의 스케일 조정을 테스트 데이터의 평균과 표준편차를 이용해서 진행하면 위와 같이 원본의 데이터 분포와 변환 후의 데이터 분포에 미세한 차이가 생긴다. 이것은 훈련세트와 테스트세트가 각각 다른 비율로 변환되었기 때문이다. 테스트 세트의 스케일이 훈련세트와 다른 비율로 조정되면 모델에 적용된 알고리즘들이 테스트 세트의 샘플 데이터를 잘못 인식한다. 그래서 테스트 셋의 표준화에도 훈련세트의 평균과 표준편차를 사용하여 스케일을 조정한다.
x_test_scaled = (x_test - train_mean) / train_std
plt.plot(x_train[:50,0], x_train_scaled[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test_scaled[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()

훈련 세트의 평균과 표준편차를 이용해서 다시 산점도를 그렸더니 원본 데이터셋의 산점도와 동일하게 변환된 것을 확인할 수 있다.
그럼 다시 한번 성능평가를 해보자.
layer2.score(x_test_scaled, y_test)
# 0.9649122807017544
스케일 조정을 진행했더니 정확도가 0.92정도에서 0.96정도로 상승했다. 이렇게 경사하강법을 사용한 모델에서는 스케일 조정만으로도 모델의 정확도를 높일 수 있다.
전체 소스 코드
from sklearn.datasets import load_breast_cancer
from sklearn.model_selection import train_test_split
cancer = load_breast_cancer()
x = cancer.data
y = cancer.target
x_train, x_test, y_train, y_test = train_test_split(x, y, stratify=y, test_size=0.2, random_state=42)
import matplotlib.pyplot as plt
print(cancer.feature_names[[2,3]])
plt.boxplot(x_train[:,2:4])
plt.xlabel('feature')
plt.ylabel('value')
plt.show()
import numpy as np
class SingleLayer :
def __init__(self) :
self.w = None
self.b = None
self.losses = []
self.w_history = []
# (1) 직선방정식 계산
def linear(self, x) :
z = np.sum(x * self.w) + self.b
return z
# (2) 활성화함수 계산
def activation(self, z) :
a = 1 / (1 + np.exp(-z))
return a
# (3) 오류역전파 계산
def backprop(self, x, err) :
w_grad = x * err
b_grad = 1 * err
return w_grad, b_grad
# (4) 훈련메소드
def fit(self, x, y, epochs=100) :
self.w = np.ones(x.shape[1])
self.b = 0
self.w_history.append(self.w.copy()) # 초기 가중치 기록
np.random.seed(42)
for i in range(epochs) :
loss = 0
indexes = np.random.permutation(np.arange(len(x)))
for i in indexes :
z = self.linear(x[i])
a = self.activation(z)
err = -(y[i] - a)
w_grad, b_grad = self.backprop(x[i], err)
self.w -= w_grad
self.b -= b_grad
self.w_history.append(self.w.copy()) # 학습 가중치 기록
a = np.clip(a, 1e-10, 1-1e-10)
loss += -(y[i]*np.log(a) + (1-y[i])*np.log(1-a))
self.losses.append(loss/len(y))
# (5) 예측메소드
def predict(self, x) :
z = [self.linear(x_i) for x_i in x]
return np.array(z) > 0
def score(self, x, y) :
return np.mean(self.predict(x) == y)
layer = SingleLayer()
layer.fit(x_train, y_train)
layer.score(x_test, y_test)
w2 = []
w3 = []
for w in layer.w_history :
w2.append(w[2])
w3.append(w[3])
plt.plot(w2,w3)
plt.plot(w2[-1], w3[-1], 'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
train_mean = np.mean(x_train, axis=0)
train_std = np.std(x_train, axis=0)
x_train_scaled = (x_train - train_mean) / train_std
layer2 = SingleLayer()
layer2.fit(x_train_scaled, y_train)
w2 = []
w3 = []
for w in layer2.w_history :
w2.append(w[2])
w3.append(w[3])
plt.plot(w2,w3)
plt.plot(w2[-1], w3[-1],'ro')
plt.xlabel('w[2]')
plt.ylabel('w[3]')
plt.show()
plt.plot(x_train[:50,0], x_train[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()
test_mean = np.mean(x_test, axis=0)
test_std = np.std(x_test, axis=0)
x_test_scaled = (x_test-test_mean)/test_std
plt.plot(x_train[:50,0], x_train_scaled[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test_scaled[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()
x_test_scaled = (x_test - train_mean) / train_std
plt.plot(x_train[:50,0], x_train_scaled[:50,1], 'bo')
plt.plot(x_test[:50,0], x_test_scaled[:50,1], 'ro')
plt.xlabel('feature1')
plt.ylabel('feature2')
plt.legend(['train_set', 'test_set'])
plt.show()
layer2.score(x_test_scaled, y_test)
[Reference.]
도서
- Do it! 정직하게 코딩하며 배우는 딥러닝 입문, 박해선 지음, 이지스퍼블리싱
[Recommended Post.]
2020/07/27 - [Deep Learning/[Books] Do it! 정직하게 코딩하며 배우는 딥러닝 입문] - [모델 구축] 이진분류 로지스틱 회귀모델 구현하기 - 심화
2020/07/23 - [Deep Learning/[Books] Do it! 정직하게 코딩하며 배우는 딥러닝 입문] - [모델 구축] 이진분류 로지스틱 회귀모델 구현하기 - 기본
2020/07/13 - [Deep Learning/[Books] Do it! 정직하게 코딩하며 배우는 딥러닝 입문] - [모델 구축] 로지스틱 손실함수와 오류 역전파 이해하기
'Deep Learning > [Books] Do it! 정직하게 코딩하며 배우는 딥러닝 입문' 카테고리의 다른 글
| [모델 튜닝] 하는 방법 - 과대적합과 과소적합 (6) | 2020.09.24 |
|---|---|
| [모델 튜닝] 머신러닝/딥러닝 Validation Set 만드는 이유와 방법 (0) | 2020.09.22 |
| [모델 구축] 이진분류 로지스틱 회귀모델 구현하기 - 심화 (0) | 2020.07.27 |
| [모델 구축] 이진분류 로지스틱 회귀모델 구현하기 - 기본 (2) | 2020.07.23 |
| [모델 평가] 훈련데이터셋 나누기 (feat.train_test_split()) (5) | 2020.07.20 |